En la actualidad, los grandes avances en ingeniería genética y biología molecular han provocado que, a nivel agrícola y de investigación científica, estemos viviendo la llamada “Era Biotecnológica” o una segunda “Revolución Verde”. La generación de organismos transgénicos mediante el uso de técnicas que posibilitan la edición génica se han convertido en potentes herramientas que sin lugar a duda han acrecentado el ritmo al que se progresa en el estudio de las plantas y sus genes.
Sin embargo, no siempre es posible emplear este tipo de procedimientos. Existen casos en los que, en el organismo de estudio en cuestión, no están bien establecidas las técnicas o los protocolos que lo permiten. En otros casos, el obstáculo viene impuesto por los propios atributos biológicos y genéticos de este, como el ciclo de vida, el tamaño del genoma o que se disponga o no de su secuencia génica. El estudio de genes que no son considerados como “modelos” de investigación puede traer consigo este tipo de inconvenientes. Aunque son evidentes las ventajas que ofrecen estas poderosas herramientas, cabe destacar algunas de las alternativas tradicionales (y totalmente complementarias) para el estudio de los genes y sus funciones.
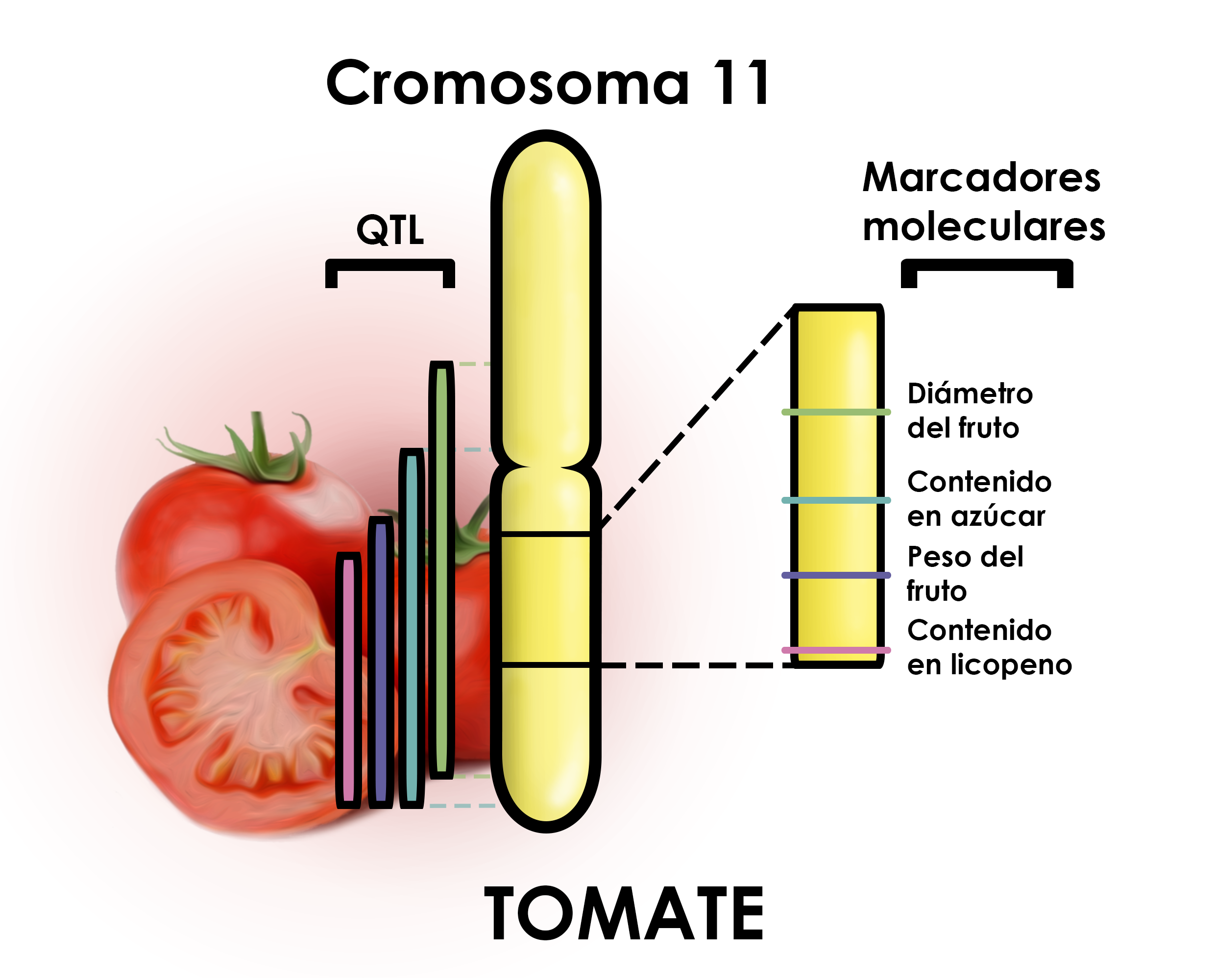
Mapa de varios QTL de interés en el cromosoma 11 del tomate. Información extraída de la base de datos de Sol Genomics Network (SGN).
Una de ellas sería el estudio de los loci de rasgos cuantitativos (QTL, Quantitative Trait Loci) a través de métodos tradicionales de selección. Un QTL es una región de ADN que está asociada con un rasgo fenotípico particular, variable y cuantificable en mayor o menor medida y que se corresponde con el producto de varios genes y el entorno. Entre sus aplicaciones está la identificación de posibles genes asociados a un rasgo fenotípico concreto, como el tamaño de las plantas o sus frutos, su sabor y color, o la resistencia a enfermedades y condiciones adversas, entre otras características. Una vez identificada dicha región de su genoma se procede a su secuenciación, lo que posibilita la comparación a través de bases de datos con genes cuya función ya está descrita. A su vez, este sistema suele ser habitual en la mejora de cultivos asistida por marcadores, también denominada Selección Asistida por Marcadores (SAM). La SAM, mediante técnicas moleculares, facilita el proceso de selección de plantas con los caracteres deseados a través de la detección de secuencias concretas de ADN ligadas al rasgo de interés. Lo interesante es que, con una simple prueba, es posible detectar en la descendencia de un cruce los individuos que presenten ese marcador y que, por tanto, hayan heredado dicho rasgo. Esto abre la posibilidad de generar variedades “élite” de plantas, donde en un mismo cultivar se han combinado varios QTL de interés, o “líneas de introgresión” que presentan un QTL concreto en homocigosis para facilitar el cruzamiento con otra planta. De esta forma, se han obtenido infinidad de cultivares y variedades de plantas de cultivo “a la carta” que están disponibles en bancos de líneas de plantas, la cuales muestran una serie de rasgos que mejoran tanto la calidad y atractividad para el consumidor como la resistencia a condiciones adversas y plagas, las cuales son una buena alternativa al uso de organismos transgénicos en la investigación.
A gran escala, es factible el análisis de un alto número de genomas o de ciertas regiones de éste para asociar las distintas variaciones genéticas específicas con ciertos QTL. Este sistema se conoce como estudio de asociación del genoma completo (GWAS por sus siglas en inglés Genome-wide association study). Tras identificar los marcadores genéticos implicados, podemos utilizarlos y entender cómo los genes involucrados contribuyen a la manifestación del rasgo en cuestión. Así, los estudios GWAS suponen una de las principales herramientas para la localización genómica de QTL y la consiguiente construcción de mapas donde se especifica, sobre los distintos cromosomas, dónde se encuentra cada uno de ellos y el marcador asociado.
La aplicación en plantas de cultivo de estos sistemas de análisis ha traído consigo un gran avance en el conocimiento de gran variedad de procesos biológicos, entre los que destaca la absorción de nutrientes del suelo. Como las raíces son el punto principal de entrada de nutrientes en las plantas, también son las que más sufren en condiciones extremas (como la sequía), las cuales pueden causar así limitaciones en la absorción mineral Es por ello que, considerando el contexto actual del cambio climático y la demanda creciente de agua y nutrientes en la agricultura intensiva, la mejora en su capacidad de absorción a través de las raíces podría aumentar la tolerancia a situaciones de estrés hídrico nutricional en los cultivos, llevando a una agricultura más eficiente. La planta Arabidopsis thaliana es el principal organismo modelo para realizar este tipo de estudios debido a la gran cantidad y a la alta calidad de datos de fenotipo y genotipo de los que se dispone, aunque igualmente se han aplicado estas herramientas en especies agrícolas como el arroz, el trigo o el tomate, por ser también organismos adecuados para el estudio de los QTL.
Por ello, un ejemplo útil es el uso de GWAS para la identificación de genes candidatos en QTL involucrados en la regulación del crecimiento de la raíz en etapa temprana y bajo diferente disponibilidad de agua y nutrientes. Es así como, por ejemplo, usando diferentes ecotipos de Arabidopsis, se han descubierto tres nuevos genes implicados en la regulación del crecimiento de la raíz bajo estrés nutricional por deficiencia de hierro y fósforo: VIM1, FH6 y VDAC3. Esto además demuestra la existencia de líneas dentro de Arabidopsis que son mejores representantes que la línea silvestre de estudio (Col-0) y alternativas al uso de OMG, por su capacidad de resistencia a la deficiencia de estos nutrientes.
Un tema actual de gran interés en el mundo de la agricultura es la producción de alimentos de alta calidad, la cual puede verse afectada, entre otros motivos, por la reducción de agua y nutrientes importantes en los suelos y por el rendimiento de los cultivos. Ambos problemas suelen venir provocados por uno mayor a nivel global como es el cambio climático. Siendo un organismo modelo representativo de plantas que generan fruto, el tomate puede considerarse uno de los más adecuados e importantes, debido a la gran cantidad de datos y resultados disponibles derivados de su investigación.
Los numerosos estudios de mapeo QTL en tomate durante los últimos años, han proporcionado gran información sobre la arquitectura genética de rasgos complejos como la dominancia y los efectos epistáticos en genes, además de su localización. Un ejemplo de esto lo podemos encontrar en el tomate doméstico (Solanum lycopersicum L.), el desarrollo de variedades resistentes al calor y a la sequía han permitido obtener un rendimiento mayor en la maduración del fruto, y por ende una mejor calidad. En relación a ello, recientes estudios con líneas individuales de la población MAGIC permitieron la localización de hasta cuatro genes candidatos de dicha función, que podrían actuar además como marcadores moleculares de mejora para una mayor resistencia a sequía.
Explicadas algunas de las ventajas y beneficios del uso de organismos transgénicos en la investigación de genes, y considerando las restricciones éticas y políticas de su uso en la agricultura mundial, se ha pretendido dar otro punto de vista del tema al destacar un conjunto de herramientas más tradicionales, pero a su vez punteras, como alternativas y complementarias al uso de estas líneas modificadas. Como se ha comentado anteriormente, estos procedimientos de estudio pueden ganar mucha importancia en función del desconocimiento que tengamos a cerca de la especie a investigar en cuestión, por lo que son vitales para dar los primeros pasos en la búsqueda de la función de sus genes. Sin embargo, aún queda mucho camino por recorrer en el estudio y caracterización de genes en plantas a través de los QTL para seguir mejorando la producción y calidad de los cultivos.